
发表期刊:the plant journal
影响因子:7.091
发表时间:2022年8月25日
研究方法:m5C merip seq、5mC medip seq、全转录组测序
文章链接:DNA and coding/non-coding RNA methylation analysis provide insights into tomato fruit ripening
云序生物提供的服务:


技术路线
1.Alisa Craig(AC)番茄果实成熟过程中的多组学特征分析
全转录组测序结果发现番茄果实中有2851个基因存在差异表达。其中1148个基因表达上调,1703个基因表达下调。由于DNA甲基化状态是番茄果实成熟调控的一个指标,随后,作者利用5mC medip seq检测了在发育过程中与番茄成熟基因相关的DNA(5mC)甲基化的变化,KEGG通路分析显示,与苯丙氨酸代谢、脂肪酸代谢、苯丙素生物合成和植物激素信号转导相关的基因被富集(图1a),GO富集分析发现,所有的差异甲基化基因都在脂质代谢过程中富集,并且在启动子、上游区域、内含子、外显子和基因间区域中都能检测到内部5mC甲基化峰,启动子上的5mC峰最多。(图1b)。结果表明,5mC在番茄基因组的所有染色体上广泛分布,低甲基化区域多于高甲基化区域,果实成熟过程中DNA(5mC)甲基化呈整体减少趋势(图1c)。此外,差异甲基化区域(DMRs)和差异表达基因(DEGs)数据的整合揭示了参与植物激素生物合成和信号转导的基因,如生长素响应蛋白IAA17(IAA17)、aba响应元件结合因子(AREB1)、生长素响应蛋白SAUR50(SAUR50)和gibbberrellin2-双加氧酶3。
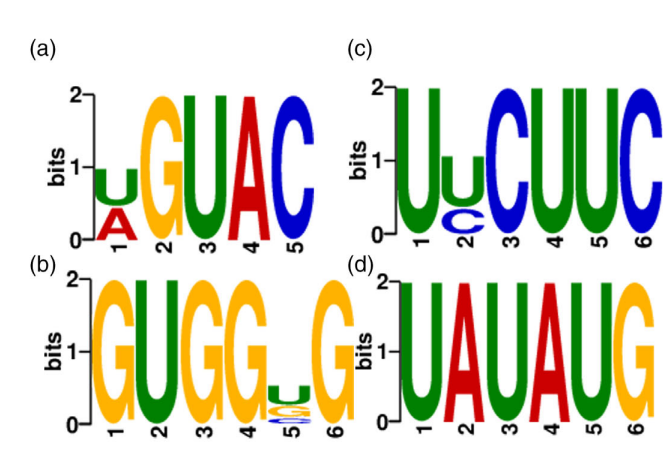
为了确定m5C峰在番茄中的分布,作者比较了AC-MG和ACBr + 6期的DMRs。m5C merip seq发现番茄中存在m5C峰富集的序列基序,如UGUAC和UUCU,此外,还发现了另外两个基序GUGGRG和UAUAUG(图2a-d)。
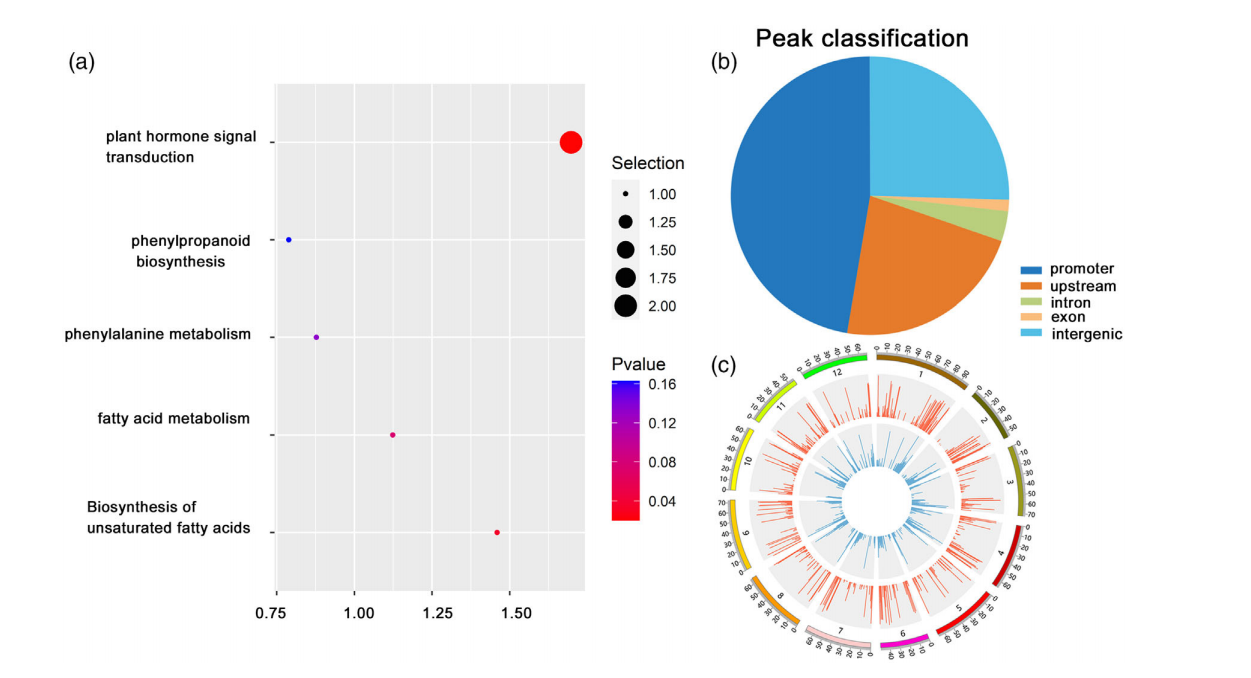
图1.绿熟期(MG)和红熟6期(Br + 6)AC番茄DNA上的5mC甲基化检测
2、番茄果实成熟过程中的多组学特征分析
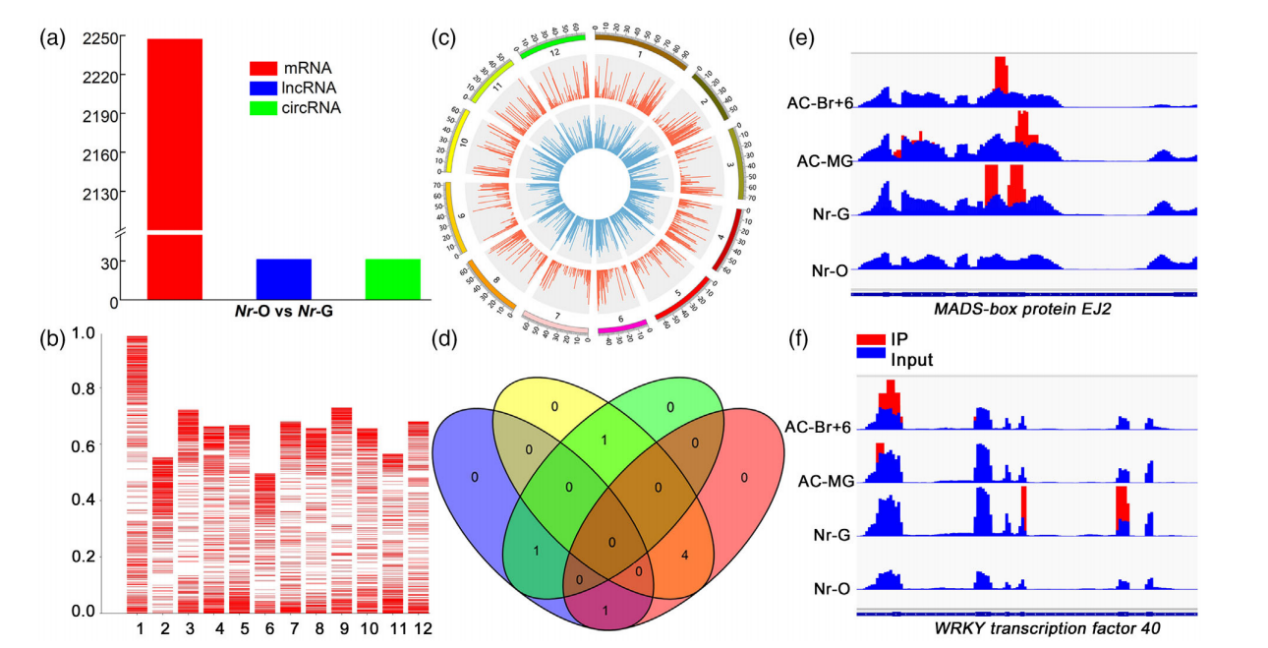
对绿熟期与黄熟期Nr果实的全转录组测序分析发现,共有2247个差异表达基因(DEGs),其中858个表达上调,1389个表达下调(图3a)。并且在Nr果实成熟过程中鉴定出了31个环状RNA和32个lncRNA的差异表达情况。此外,作者还进行了DNA(5mC)甲基化分析,以深入探究了Nr突变体果实在不同阶段的(5mC)水平。在基因组水平上,DMRs在每个染色体上的分布并不均匀;1号染色体甲基化区域最多,6号染色体甲基化区域最少(图3b),在12条染色体上,共鉴定出1694个上调的DMRs和4190个下调的DMRs(图3c)。研究结果表明,RNA甲基化(m5C)能功能调控基因的表达,但是m5C与mRNA丰度之间的相关性仍有待进一步探索(图3d)。此后,为了验证m5C丰度的差异,作者选择了参与果实成熟的基因,并验证了WRKY TFs和MADS-box蛋白EJ2中m5C的丰度变化(图3e-f)。
3、AC和Nr果实绿熟期的多组学概况分析
对AC和Nr果实成熟绿色期的DEG进行比较分析,共发现1981个差异表达基因,其中977个表达上调,1004个表达下调(图4a-b)。在AC和Nr突变体果实之间共鉴定出100个DMRs,其中许多都参与了果实的成熟过程,并且发现5mC在基因间和上游DNA区域周围高度富集(图4c)。此外,KEGG分析表明,甲基化基因富集于萜类主干生物合成、淀粉和蔗糖代谢以及戊糖和葡萄糖醛酸盐的相互转化的通路上(图4d),随后,作者研究了Nr果实成熟过程中与基因表达相关的乙烯释放过程中DNA甲基化(5mC)的变化,发现EBF1和ERF6被高甲基化,同时Nr-G和AC-MG的表达表达下调(图4e)。一些DMRs和DEGs,如a-Man、PG、CESA2、PEI、PE和XTH23也被发现参与了果实的质地调控(图4f)。
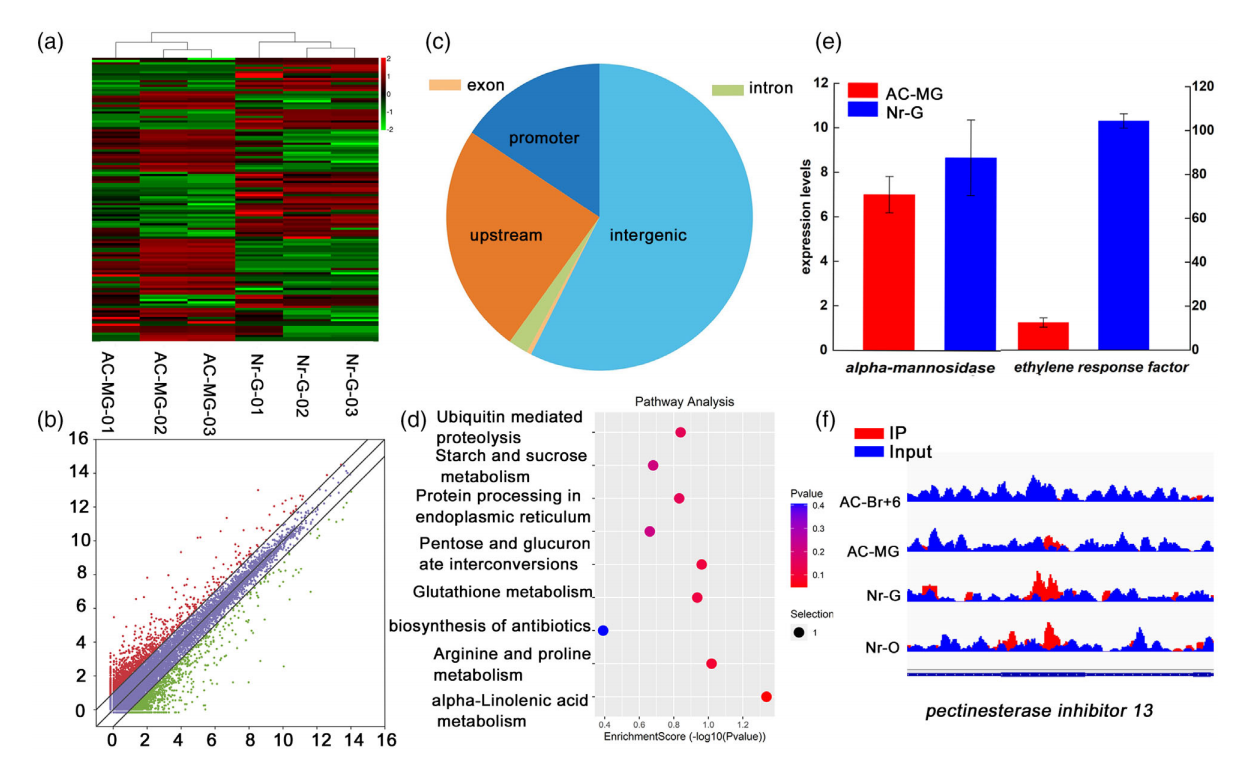
4、AC与Nr果实成熟期的多组学谱分析
作者比较了Nr-O和AC-Br + 6果实之间的DEGs,发现了2261个DEGs,其中1190个表达上调,1071个表达下调(图5a)。共发现21个差异表达(DE)的 lncRNA和33个DE环状RNA,其中有11个lncRNA和16个环状RNA显著上调,而10个lncRNA和17个环状RNA被下调(图5a)。DMRs的GO和KEGG通路分析显示,Nr-O和Nr-G中存在许多与油菜素内酯生物合成、倍半萜生物合成、三萜生物合成、二萜生物合成和类胡萝卜素生物合成相关的基因。根据MeDIP-seq的结果,这些序列的长度在1000~10000bp之间(图5b),此外,作者还发现了许多编码转录因子的基因,如bHLH18/74/90/123、MYB13/20/21/36/41S3和WRKY49/72/ 77(图5c)。
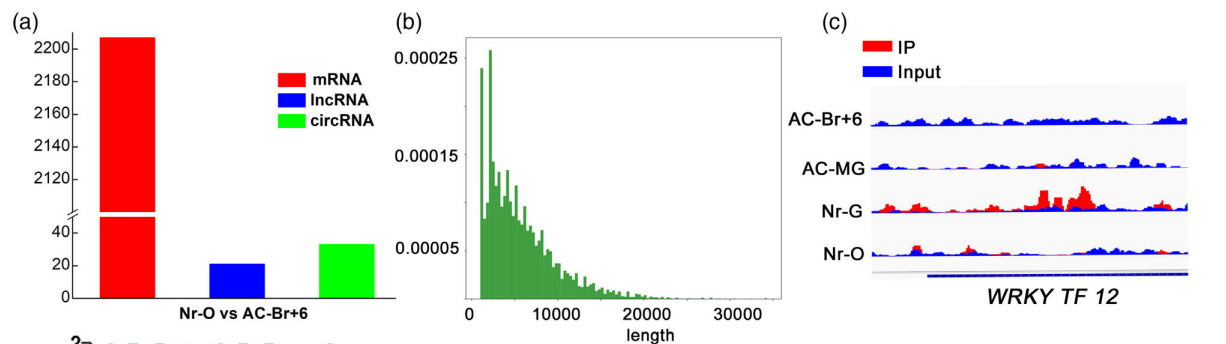
5.AC和Nr突变体果实成熟过程的多组学比较分析
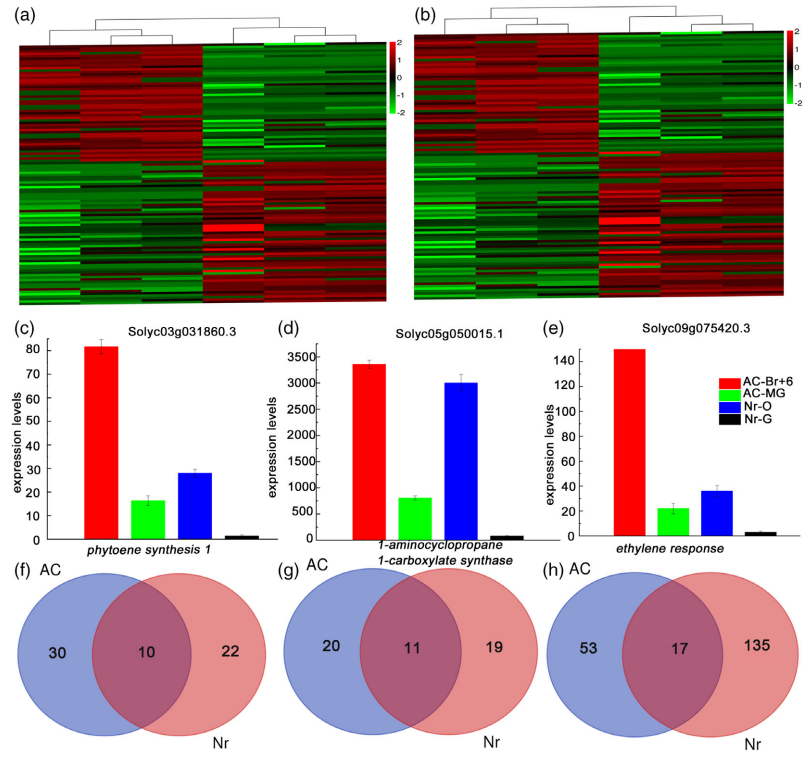
为了确定AC果实和Nr突变体果实成熟过程之间的差异,作者比较了AC-MG与AC- Br + 6和Nr-MG与Nr-O之间的DEGs,共发现63个DEGs,其中14个表达上调,49个表达下调(图6a)。相比之下,仅Nr突变体的果实中就有86个与成熟相关的基因差异表达,其中33个基因表达上调,53个基因表达下调(图6b)。随后,通过比较AC和Nr果实中与细胞壁代谢相关的基因,作者发现这些基因在Nr中的表达倍数变化大于AC果实。并且乙烯合成和信号转导相关基因的表达量Nr果实大于AC果实。为了进一步探究果实间的成熟过程差异,作者还分析了Nr-O、Nr-G、ACBr + 6和AC-MG组的环状RNA、lncRNAs及其相应的靶基因。在AC和Nr果实中发现11个lncRNA和10个环状RNA为差异表达,其中3个lncRNA上调,8个lncRNA下调,6个环状RNA上调,4个环状RNA下调,并且在Nr中特异性鉴定出19个lncRNA和22个环状RNA(图6f-g)。通过DMRs和DEGs联合分析结果的维恩图,显示了AC-MG和AC-Br + 6与Nr-MG和Nr-O对照组中常见和特异性的甲基化基因,并发现在Nr突变体中表达的特定基因中,96个DEGs表达降低,39个表达增加(图6h)。
该篇文章联合m5C merip seq和5mC medip seq技术,在转录和表观遗传水平上探讨了肉质果实成熟过程中的调控关系,并构建了协调调控网络模型(图7),发现AC和Nr中的DMRs和DEGs协同作用与乙烯释放、类胡萝卜素积累和细胞壁代谢有关,从而调节果实成熟。首次揭示了mRNA和ncRNA m5C的表达谱,为今后研究果实的成熟和衰老提供了参考数据。
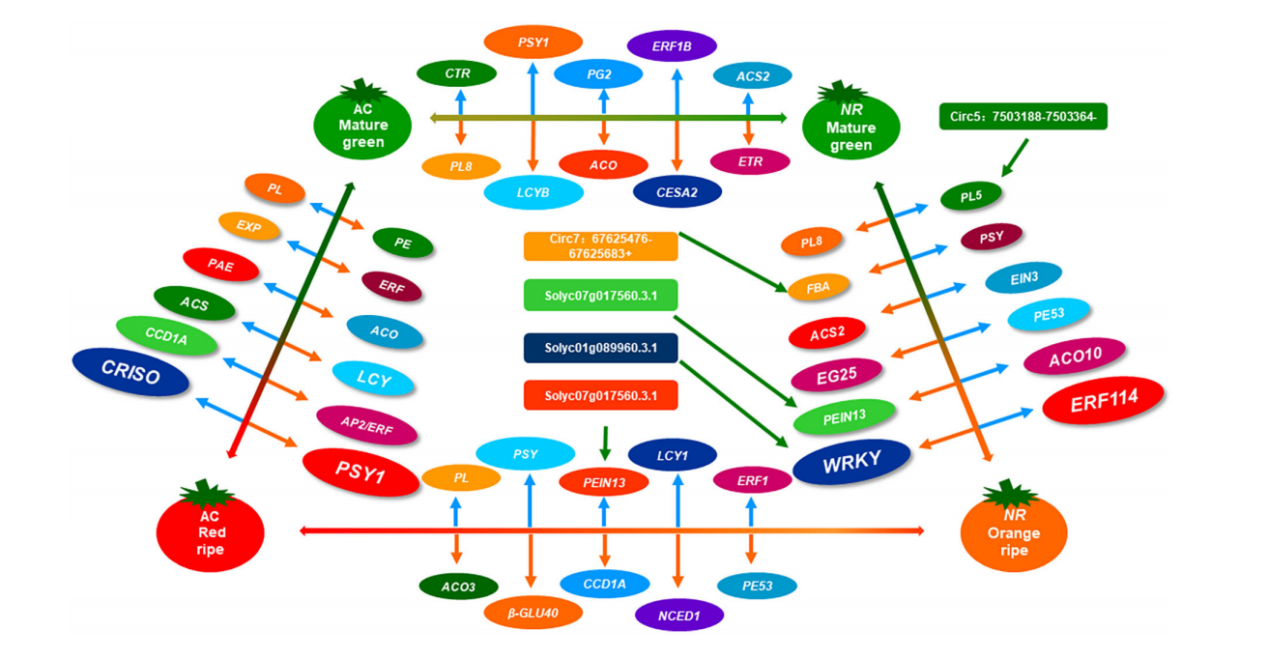
图7.果实成熟过程中乙烯反应和类胡萝卜素积累、细胞壁分裂和芳香化合物相关途径的调控模型。
云序生物m5C甲基化研究五大模块
01 m5C RNA甲基化测序
对m5C RNA甲基化,目前最流行的检测手段为m5C-Seq技术,适用于m5C RNA甲基化谱研究,快速筛选m5C RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m5C测序:
m5C 全转录组测序(涵盖mRNA,LncRNA,circRNA)
m5C LncRNA测序(涵盖LncRNA和mRNA)
m5C Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
m5C mRNA测序
02 检测整体m5C RNA甲基化水平
精准高效,可以实现一次检测,9类修饰水平检测,一步到位。
03 m5C RNA甲基化上游酶的筛选
m5C RNA甲基化相关酶PCR芯片
寻找上游直接调控m5C RNA甲基化的甲基转移酶。
04 m5C RNA甲基化靶基因验证
云序提供各类不同修饰的meRIP-qPCR服务,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
05 机制互作研究
5.1 RIP-seq/qPCR
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
5.3 双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
5.4 ChIP-seq
筛选或验证目标蛋白与DNA互作,研究相应的分子调控机制。
DNA甲基化:DNA甲基化能控制基因表达,因而在动植物的生长发育过程中发挥着关键的调控作用,并且与包括癌症在内的多种人类疾病密切相关,是表观遗传学研究的热点。
01 MeDIP甲基化测序
将甲基化DNA免疫共沉淀技术(MeDIP)和高通量测序相结合,能够帮助客户快速地获取全基因组范围内的DNA甲基化图谱,并比较不同样品中的甲基化分布差异。配合强大的生信分析实力,我们提供的不仅是海量的测序数据,而且是根据您的研究目的,有针对性地挖掘提炼出取之即可用的结果及出版级图片。
MeDIP优势:
1.1 相比于WGBS全基因组检测,性价比更高!
1.2实现对甲基化(5mC)和羟甲基化(5hmC)的区分。
02 ctDNA EM-seq甲基化测序
针对体液样品中的 ctDNA 进行优化,云序生物为您带来一种基于酶学方法的 DNA甲基化测序技术 EM-seq(Enzymatic Methyl-seq)。该方法结合使用多种酶,在保证 5mC 和 5hmC 在 Illumina 测序中仍被识别为 C 的前提条件下,将未发生修饰的胞嘧啶(C)转化为尿嘧啶(U),并在后续的扩增和测序过程中被识别为胸腺嘧啶(T)。EM-seq 有效弥补了全基因组重亚硫酸盐法测序(WGBS)需要极端反应条件的缺陷,可以广泛应用于包含 ctDNA 在内的微量脆弱 DNA 样品。运用 EM-seq 技术,仅需低至 10 ng 的 DNA 样品便可进行单碱基精度的全基因组 5mC 修饰测序。
EM-seq 的优势相比于传统的WGBS 拥有多种优势:
2.1 DNA 测序文库质量高、所需 DNA 起始量小
相比于WGBS,EM-seq 对 DNA 的损伤小,因此可以用更少 DNA 样品、更少的 PCR 轮数扩增出高质量的测序文库。云序生物提供的 EM-seq 服务,所需的 DNA 起始量可低至10ng。
2.2 DNA 文库的插入片段更长、更完整
相比于WGBS,EM-seq 处理后的 DNA 片段更完整,长度更长,避免产生测序缺口。
2.3 出色的 GC 覆盖均一性
无论 GC 程度高低,EM-seq 的覆盖度都有均匀且优良的表现;相比之下,WGBS 测序文库高估了 AT 区,却低估了 GC 区,造成“GC 覆盖度偏误”。
2.4 均一的双核苷酸分布
由于“GC 覆盖度偏误”的存在,在连续双核苷酸的检出效率方面,WGBS 对于不同的双核苷酸存在较严重的偏误,而 EM-seq 则能展示出均一的覆盖情况。
2.5 更强的匹配率和更低的重复率
相比于 WGBS,EM-seq 测序结果与参考基因组的匹配率更高,同时重复率更低。
2.6 用更少的测序检出更多的 CpG 岛
同等测序覆盖深度的情形下,EM-seq 所发现的 CpG 数目要远多于 WGBS,在这其中有很大比例的 CpG 位点是仅仅只能能用 EM-seq 法才能检测到的。
优势一:云序累计支持客户发表 87 +篇RNA修饰及DNA修饰SCI论文,合计影响因子 810 +
优势二:累计完成数千例 RNA及DNA甲基化测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各
